Projekte
Sympore - Plasmodesmata as Symplasmic Pores for Plant Cell-to-Cell Communication

Hohenheimer Online Kurier 2020-11-11
AMAIZE-P: Adaptation of maize-based food-feed-energy systesm to limited phosphate resources

Wir untersuchen die proteinregulatorischen Netzwerke, die in verschiedenen Maisgenotypen in Antwort auf niedrigen Phosphatverfügbarkeit induziert werden. Wir führen eine Kombination von Labor- und Feldversuchen durch.
RS 1.3 Regulatory modules of carbon resource allocation under different phosphate availabilities
Arabidopsis Kinom und Proteinphosphorylierung in Pflanzen
Kinase - Target Interaktionen und Proteinphosphorylierungsstellen in Pflanzen: PhosPhAt
Wir interessieren uns für proteomweite Proteinphosphorylierung und die zugehörigen Kinasen. In der Datenbank "PhosPhAt" haben wir umfassende information über Proteinphosphorylierung in Arabidopsis aus zahlreichen large-scale Phosphoproteomics-Experimenten zusammengetragen.
In den letzten Monaten wurde die Datenbank erweitert um regulatorische Informationen zu Proteinkinasen und ihren Targets. Nach intensiver Literaturarbeit konnten detaillierte Informationen zu etwa 400 Proteinkinasen gefunden werden und in die Datenbank eingepflegt werden.
Phosphorylierungsdynamik und regulatorische Netzwerke
Die pflanzliche Plasmamembran ist ein dynamisches Kompartiment, das wichtig ist für den Transport von Nährstoffen und Metaboliten, sowie der Begrenzung von Zellen und zur Verankerung der Zellwand. Eine weitere wichtige Funktion der Proteine und Lipide in der Plasmamembran ist die Kommunikation von Zellen mit der Umgebung. Es sind daher zahlreiche Proteine in die Membran eingelagert, die für Aufnahme und Erkennung von Nährstoffen wichtig sind. Diese Proteine können durch Phosphorylierung aktiviert oder inaktiviert werden. Mit Hilfe quantitativer Proteinmassenspektrometrie konnten wir in der Vergangenheit erstmals die Dynamik der Proteinphosphorylierung nach Zugabe unterschiedlicher Nährstoffe abbilden. Dabei zeigte sich, dass sich die maximalen Änderungen der Proteinphosphorylierung innerhalb von 30 Minuten von den frühen Änderungen bei Membranproteinen hin zu den späteren Antworten bei löslichen Proteinen (Enzymen) verschiebt. Eine bioinformatische Analyse und Rekonstruktion von zeitabhängigen Phosporylierungsnetzwerken identifizierte einige membranständige Kinasen, die bei der Regulation der Nährstoffaufnahme eine Rolle spielen können.
Charakterisierung von Membranmikrodomänen
In Pflanzen, ebenso wie in tierischen Zellen und in Hefe, ist die Plasmamembran keine homogene "Hülle", sondern unterteilt in Bereiche mit unterschiedlicher Lipid- und Proteinzusammensetzung. Die Membran besteht überwiegend aus einer Phospholipid-Doppelschicht, in die als weitere Fettsäuren auch Sterole und Sphingolipide eingebettet sind. Die Proteine sind als Transmembranproteine in diese Lipiddoppelschicht eingefügt oder mit Hilfe von hydrophoben Modifikationen, wie zum Beispiel Palmyl-, Myristol- oder Glycosylphosphatidylinositol-resten an die Membran angeheftet.
In tierischen Zellen und in Hefe konnte bereits seit längerem gezeigt werden, dass die Sterol-Lipide sich nicht gleichmäßig in der Lipiddoppelschicht verteilen, sondern sich physikalisch von den Phospholipid-Bereichen entmischen. Es entstehen sterol-reiche Membranbereich mit höherem „Ordnungsgrad“ (lipid ordered phase), sogenannte Membranmikrodomänen oder "Lipid-Rafts". Biochemisch lassen sich diese Sterol-reichen Membrandomänen aufgrund ihrer Resistenz gegen bestimmte nicht-ionische Detergenzien über Gradientenzentrifugation aufreinigen. Die sterol-reichen Membrandomänen, die auch als "Detergenzien-resistente Membranen" bezeichnet werden, haben niedrigere Dichte als die Phospholipid-haltigen Membranbereiche und sind in einem Saccharosegradienten als charakteristische Fraktion abzunehmen, und eine Stimulus-induzierte Veränderung der Zusammensetzung dieser sterol-reichen Regionen konnte gezeigt werden.
Wir möchten die Rolle dieser Membranmikrodomänen für pflanzliche Anpassungen an sich ändernde externe Bedingungen untersuchen. Dazu nutzen wir Sterol-Biosynthese Mutanten, die aufgrund ihres veränderten Sterolgehaltes der Plasmamembran eine veränderte Struktur von Membranmikrodomänen besitzen. Die Änderung der Membranmikrodomänenzusammensetzung soll nach Stimulus mit unterschiedlichen Nährstoffen (Zucker, Stickstoff) untersucht werden.
Methodenentwicklung in der quantitativen Proteomik
Da wir uns vor allem für die Rolle von Proteinenn und Proteinmodifikationen in Signaltransduktionsprozessen interessieren, nutzen wir Metoden der quantitativen Proteomik. Um diese Methoden optimal an die jeweiligen biologischen Fragestellungen anzupassen, ist auch Neu- und Weiterentwicklung von analytischen Methoden wichtig.
In der Vergangenheit haben wir vor allem die metabolische Markierung mittels 15N für Pflanzen etabliert und angewendet. Außerdem konnten wir Verbesserungen und Automatisierungen in der Datenverarbeitung und statistischen Analyse umsetzen (cRacker).
(beendet) FOR1061: Bedeutung von Phosphorylierung von Tonoplastenproteinen für die Aktivität und das Proteintageting
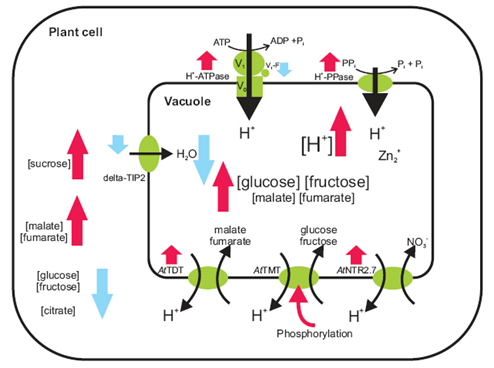 Ziel der Forschergruppe FOR1061 ist die Analyse von Vakuolenfunktionen mit Fokus auf regulatorische Wechselwirkungen an Tonoplastenproteinen bei Stressanpassung (Kälte und Trockenstress). Im Rahmen der Forschergruppe FOR1061 ist die Aufgabe des Fachgebiets "Systembiologie der Pflanze" der Aufbau (i) einer gerichteten Analytikplattform mit Hilfe von „Selected Reaction Monitoring“ und unter Verwendung von mit stabilen Isotopen markierten, aneinandergehängten Standardpeptiden (QconCAT), sowie (ii) die gezielte Untersuchung des Phosphorylierungsstatus von Tonoplastentransportern der TMT und ERD-Familie in Zusammenhang mit Transportaktivität. Schließlich ist (iii) die unterichtete Analyse von Proteintargeting an Plasmamembran und Tonoplast in Mutanten des Proteintargeting-Pathways (z.b. ap3-Mutante) geplant. Hierfür soll ein proteomischer Ansatz unter Verwendung von 15N-metabolischer Markierung benutzt werden und die Fraktionierung von Membranfraktionen in Tonoplasten und Plasmamembran.
Ziel der Forschergruppe FOR1061 ist die Analyse von Vakuolenfunktionen mit Fokus auf regulatorische Wechselwirkungen an Tonoplastenproteinen bei Stressanpassung (Kälte und Trockenstress). Im Rahmen der Forschergruppe FOR1061 ist die Aufgabe des Fachgebiets "Systembiologie der Pflanze" der Aufbau (i) einer gerichteten Analytikplattform mit Hilfe von „Selected Reaction Monitoring“ und unter Verwendung von mit stabilen Isotopen markierten, aneinandergehängten Standardpeptiden (QconCAT), sowie (ii) die gezielte Untersuchung des Phosphorylierungsstatus von Tonoplastentransportern der TMT und ERD-Familie in Zusammenhang mit Transportaktivität. Schließlich ist (iii) die unterichtete Analyse von Proteintargeting an Plasmamembran und Tonoplast in Mutanten des Proteintargeting-Pathways (z.b. ap3-Mutante) geplant. Hierfür soll ein proteomischer Ansatz unter Verwendung von 15N-metabolischer Markierung benutzt werden und die Fraktionierung von Membranfraktionen in Tonoplasten und Plasmamembran.
